Neues Modell für landkreisbezogene Corona-Vorhersagen - Neuroinformatiker der Universität Osnabrück und Datenspezialisten des Jülich Supercomputing Centre liefern täglich aktuelle Auswertungen des Infektionsgeschehens
OSNABRÜCK / JÜLICH.- Neuroinformatiker der Universität Osnabrück und Datenspezialisten des Forschungszentrums Jülich stellen täglich neue Modellergebnisse zur Vorhersage der Covid-19-Infektionen bereit. Die Ergebnisse enthalten tagesaktuelle Schätzungen der gemeldeten Neu-Infektionen und eine 5-Tages-Vorhersage für jeden deutschen Landkreis, und sind auf der Website https://covid19-bayesian.fz-juelich.de abrufbar.
148/2020 23.09.2020
Neues Modell für landkreisbezogene Corona-Vorhersagen -
Neuroinformatiker der Universität Osnabrück und Datenspezialisten des Jülich Supercomputing Centre liefern täglich aktuelle Auswertungen des Infektionsgeschehens
OSNABRÜCK / JÜLICH.- Neuroinformatiker der Universität Osnabrück und Datenspezialisten des Forschungszentrums Jülich stellen täglich neue Modellergebnisse zur Vorhersage der Covid-19-Infektionen bereit. Die Ergebnisse enthalten tagesaktuelle Schätzungen der gemeldeten Neu-Infektionen und eine 5-Tages-Vorhersage für jeden deutschen Landkreis, und sind auf der Website https://covid19-bayesian.fz-juelich.de abrufbar.
Die Prognosen beruhen auf Daten des Robert Koch-Instituts, die mit einem neuen, wahrscheinlichkeitsgewichteten Modell der Osnabrücker Neuroinformatiker auf Höchstleistungsrechnern des Jülich Supercomputing Centre (JSC) statistisch analysiert werden.
Das "COVID-19 Bayesian Modelling for Outbreak Detection", kurz BSTIM-Modell, besitzt zwei wesentliche Merkmale, die es von anderen Methoden unterscheidet: "Zum einen liefert die neue Methode einen Vorhersagehorizont, der es ermöglicht, die Verlässlichkeit der Prognosen zu beurteilen. Weiterhin wird der Einfluss vom örtlich benachbarten Infektionsgeschehen mit einbezogen. Das erlaubt uns, auch die Dynamik der Ausbreitung zu bewerten", erklärt Prof. Dr. Gordon Pipa, Leiter der Arbeitsgruppe Neuroinformatik auf dem KI-Campus an der Universität Osnabrück, die das BSTM-Modell erst kürzlich in einem anderen Kontext entwickelt hat.
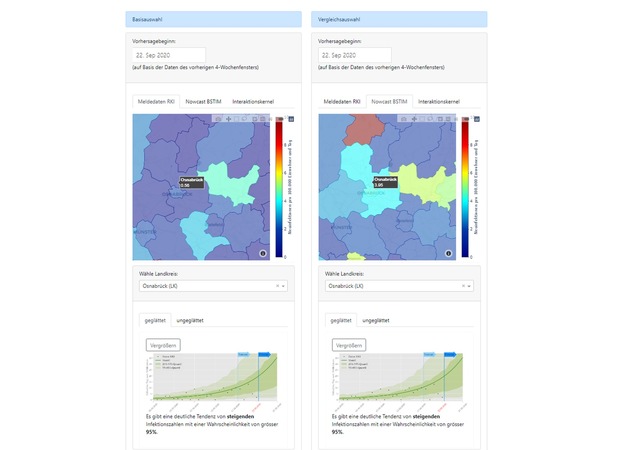
Eine von mehreren Herausforderungen bei der Auflösung nach einzelnen Landkreisen sind die niedrigen Fallzahlen. "Eine einzelne Prognosekurve kann irreführend sein, da sich die Verlässlichkeit der Vorhersage nicht beurteilen lässt", erläutert Prof. Pipa. "Das von uns verwendete BSTIM-Modell berechnet daher nicht nur einen einzelnen wahrscheinlichen Verlauf, sondern berücksichtigt viele mögliche Verläufe, die zu den Daten konform sind. Dies ermöglicht die Berechnung sogenannter Vorhersagehorizonte als Maß für die Streuung der Wahrscheinlichkeiten. Auf diese Weise ist eine Einschätzung der Situation einschließlich statistischer Unsicherheiten möglich, die auch bei niedrigen Fallzahlen hilfreiche Informationen liefern kann."
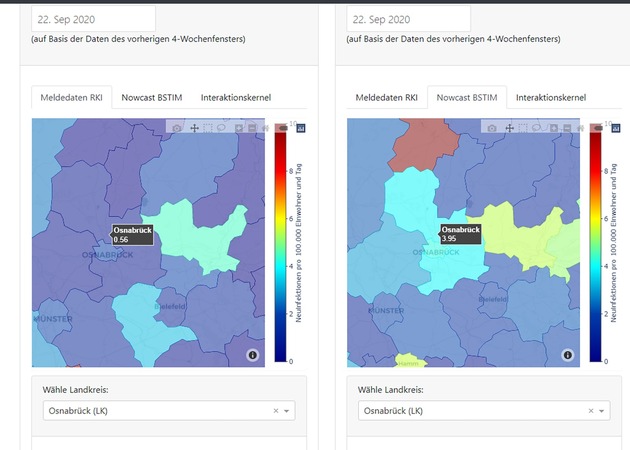
Das BSTIM-Modell errechnet überdies den Einfluss angrenzender Regionen. Ein sogenannter Interaktionskernel beschreibt, wie sehr sich eine hohe oder niedrige Infektionszahl in einer benachbarten Region auf das Infektionsgeschehen in einem Landkreis auswirkt. Die Osnabrücker Arbeitsgruppe Neuroinformatik und das Robert Koch-Institut hatten den Interaktionskernel bereits 2019 erfolgreich eingesetzt, um den Verlauf von Infektionen mit dem Rotavirus sowie Borreliose und Campylobacter-Bakterien zu beschreiben.
Experten des Jülich Supercomputing Centre (JSC) halfen dabei, die Methode für COVID-19-Daten zu adaptieren, und den Code für die Analyse auf den Jülicher Superrechnern anzupassen. "Die Ermittlung des Vorhersagehorizonts ist äußerst rechenintensiv, da wir dazu auf sehr viele Modellvariationen abgewiesen sind. Daher braucht die statische Modellierung deutlich mehr Rechenzeit als Methoden die auf einen Vorhersagehorizont verzichten. Um die Analysen täglich unmittelbar nach der Verfügbarkeit der RKI-Daten zur Verfügung stellen zu können, nutzen wir Ressourcen des Jülich Supercomputing Centre - normale Arbeitsplatzrechner wären damit weit überfordert", erklärt Jens Henrik Göbbert vom JSC.
Die tagesaktuellen Auswertungen sowie die Möglichkeit räumlicher und zeitlicher Vergleichsdarstellungen sind öffentlich zugänglich und werden auf der Webseite https://covid19-bayesian.fz-juelich.de möglichst allgemeinverständlich dargestellt. "Wir wollten die Ergebnisse schnell und auf verständliche Weise zur Verfügung stellen, damit eine breite Zielgruppe zeitnah über Inhalte reden kann und man sich nicht an der Technik aufhalten muss", erklärt Göbbert.
Besucherinnen und Besucher der Seite können etwa interaktiv 5-Tages-Prognosen für frei wählbare Landkreise aufrufen, oder die aktuellen Meldedaten des Robert-Koch-Instituts mit den geschätzten tatsächlichen Neuinfektionen vergleichen. Aufgrund von Verzögerungen bei der Datenübermittlung weichen die gemeldeten Zahlen oftmals und teils erheblich von der tatsächlichen Zahl der neuen Fälle ab. Ein sogenannter "Nowcast" verfolgt dabei das Ziel, zunächst die aktuellen Zahlen mittels statistischer Analysen abzuschätzen. Ein "Forecast" liefert eine Schätzung der Entwicklung für weitere fünf Tage.
Auch für künftige Herausforderungen im Bereich der Pandemieforschung hat das Projektteam schon eine Idee. Prof. Pipa: "Solche interdisziplinären Teams könnten als Kompetenzzentren über die Grenzen von Instituten hinweg etabliert werden, um sich zum Beispiel durch Simulation von Krisensituationen auf verschiedene Szenarien und deren notwenigen Schritte bei der Datenbeschaffung und Datenanalyse vorzubereiten, ähnlich dem Katastrophenschutz."
Aktuelle Vorhersagen der Corona-Infektionen nach Landkreisen:
https://covid19-bayesian.fz-juelich.de
Video: Wie funktioniert die Vorhersage und Analyse
Weitere Informationen:
Tagesaktuelle Berechnungen der Reproduktionszahl für deutsche Bundesländer: https://www.fz-juelich.de/portal/DE/Forschung/Rtlive/_node.html
Ansprechpartner:
Prof. Dr. Gordon Pipa, Universität Osnabrück
Institute of Cognitive Science
Wachsbleiche 27, 49090 Osnabrück
Tel. +49 541 969 2277
E-Mail: office@ni.uni-osnabrueck.de
Jens Henrik Göbbert, Forschungszentrum Jülich GmbH
Jülich Supercomputing Centre
Wilhelm-Johnen-Straße, 52425 Jülich, Germany
Tel. +49 2461 61 96498
E-Mail: j.goebbert@fz-juelich.de
Ansprechpartner für die Medien:
Dr. Utz Lederbogen
Pressesprecher der Universität Osnabrück
Neuer Graben 29, 49069 Osnabrück
Tel. +49 541 969 4370
E-Mail: utz.lederbogen@uni-osnabrueck.de
Tobias Schlößer, Forschungszentrum Jülich
Unternehmenskommunikation
Tel. +49 2461 61 4771
E-Mail: t.schloesser@fz-juelich.de
Dr. Utz Lederbogen Pressesprecher Universität Osnabrück Tel. +49 541 969 4370 E-Mail: utz.lederbogen@uni-osnabrueck.de
Weiteres Material zum Download Dokument: 148_PM_Pipa_Corona_V~ersage20200923.docx